生命科学与技术学院刘琦团队开发基于元学习的AI模型进行抗原-TCR亲和力识别,相关研究成果发表于《自然・机器智能》
来源:生命科学与技术学院
时间:2023-03-14 浏览:
T细胞受体(T-cell receptor, TCR)是获得性免疫过程中的关键分子。TCR经过特定的基因重组和进化筛选,具备高度的多样性及特异性。MHC-多肽复合物与TCR亲和力的计算识别(pMHC-TCR binding recognition)是揭示肿瘤、自身免疫性疾病和病毒感染性疾病等疾病发生发展机制的重要手段,也是计算免疫学领域基本而又极具挑战的问题,其挑战性具体体现为:(1)TCR空间呈现高度多样性,现有TCR识别的计算模型难以泛化至该高度多样化的TCR空间;(2)已知肽段-TCR配对数据服从长尾分布(Long-tail)(图1),训练数据样本分布严重不均衡,少量的肽段拥有大量已知TCR结合数据(头部肽段),但大部分肽段仅记录了少量已知的TCR结合信息(尾部肽段)。直接基于此数据构建传统的监督式学习模型会使得模型倾向于学习头部样本的TCR结合模式,但难以泛化至尾部样本(Few shot)的亲和力预测;(3)肿瘤新生抗原,外源性肽段等对于免疫系统来说是未见的新生抗原,对于该类抗原的TCR识别是免疫治疗和细胞治疗的关键。但该类抗原的TCR亲和力识别属于AI领域的零样本识别问题(Zero shot),现有的计算模型无法解决。
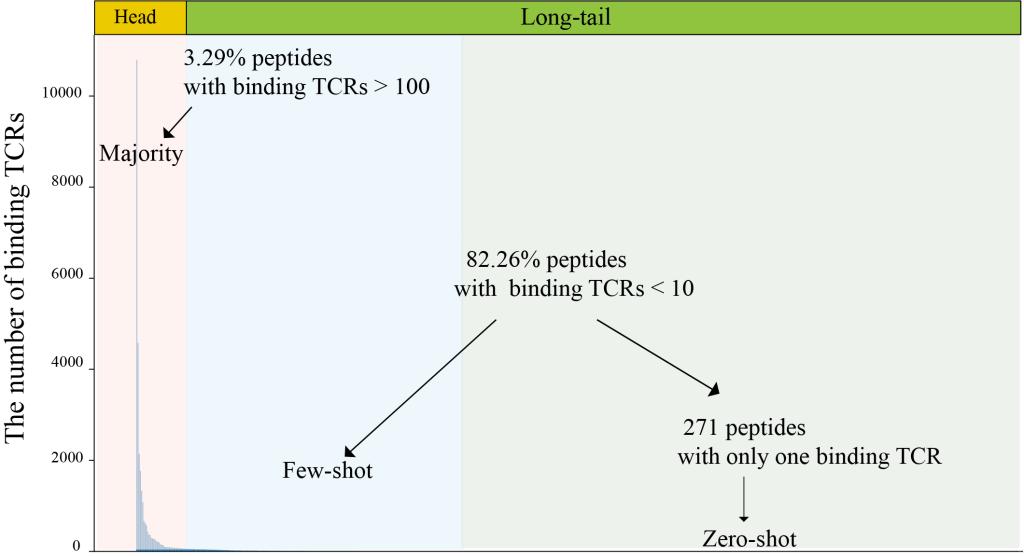
图1.已知数据肽段-TCR结合数据服从长尾分布
近日,生命科学与技术学院生物信息学系、上海自主智能无人系统科学中心刘琦教授课题组在国际人工智能领域顶级期刊《自然・机器智能》(Nature Machine Intelligence)上发表了题为“Pan-Peptide Meta Learning for T-Cell Receptor-Antigen Binding Recognition”的论文,发布了普适有效的抗原-TCR亲和力预测的AI模型PanPep。面向上述pMHC-TCR亲和力识别中的挑战和瓶颈,创新性地提出了基于元学习(Meta Learning)和神经图灵机(Neural Turning Machine)的AI计算框架,通过模拟人类对于已知任务的存储记忆和新任务的类比学习机制,有效地解决上述数据的长尾分布识别问题:即面向尾部肽段(Few shot)和肿瘤新生抗原或外源性肽段(Zero shot)进行TCR亲和力识别。实验证明PanPep在三种应用场景Majority learning,Few-shot learning以及Zero-shot learning场景中均取得了较高的抗原-TCR预测准确率。
PanPep算法框架(图2)包含了元学习模块和解耦蒸馏(Disentanglement distillation)模块。其中,针对已知数据的长尾效应,元学习模块采用了Model-Agnostic Meta Learning(MAML)计算框架。模型假设每一个肽段具有其特异性的TCR结合模式,因此每一个肽段下的TCR结合识别任务被当作MAML中的一个任务,且肽段表征的分布即为任务的分布。基于这些肽段任务(Peptide-specific task),元学习模块能够仅基于少量训练样本在不同任务中快速泛化。而对于诸多未见的肽段,例如新生抗原、外源性抗原等肽段,由于缺乏已知的TCR结合信息,无法通过对于元学习模块进行微调使其快速泛化到该任务上,故研究团队受启发于神经图灵机(NTM)通过外部记忆模块避免学习遗忘这一机制,创新性地开发了解耦蒸馏模块,借助以往学习任务所获得的经验,对于未见的新肽段的TCR结合识别任务进行泛化,从而实现零样本学习。
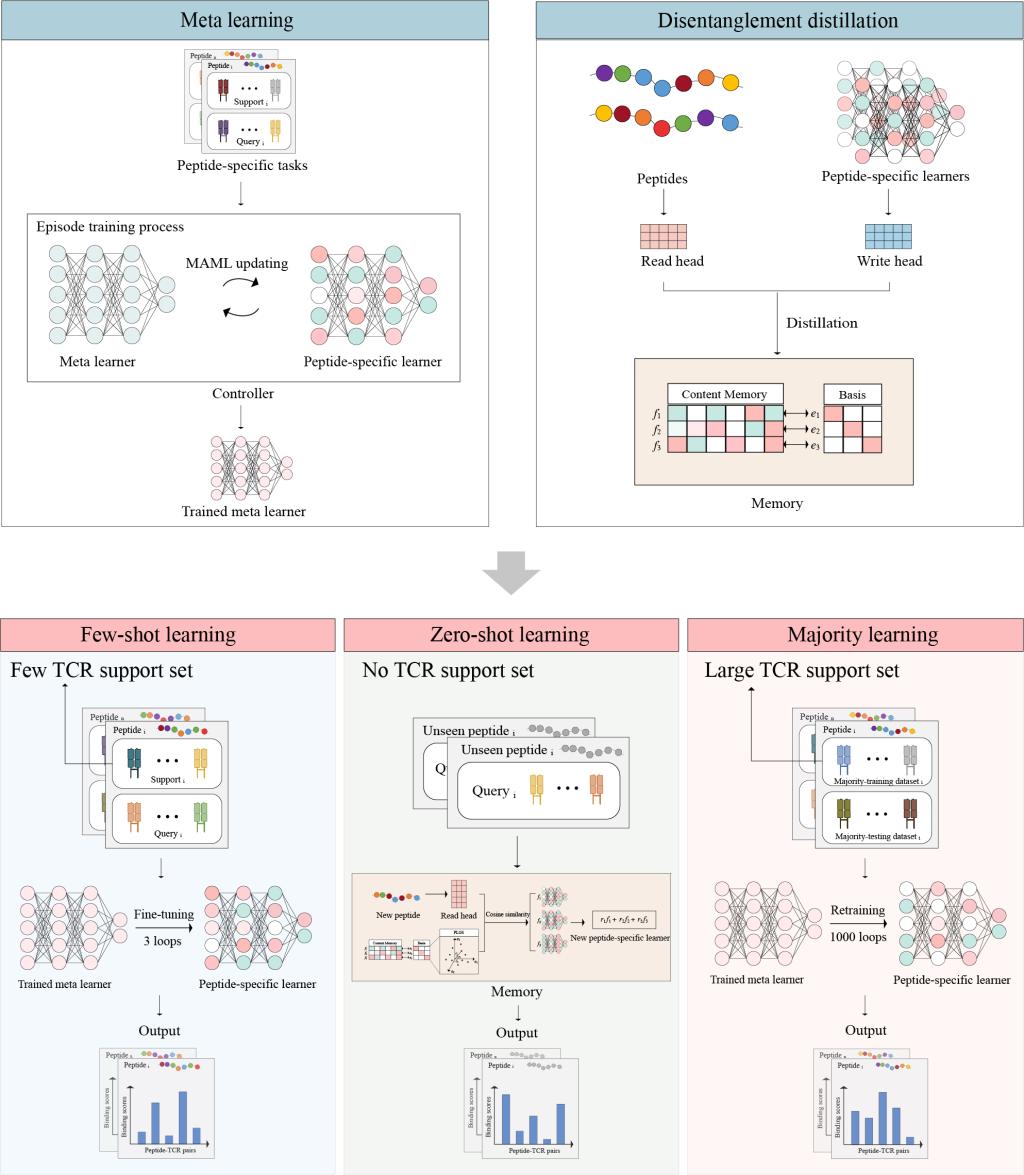
图2.PanPep算法框架
该工作中,研究团队首先将该算法与同类算法在三种测试场景(Majority,Few-shot和Zero-shot场景)中进行比较,PanPep在保持Majority场景的预测性能的同时,在Few-shot和Zero-shot场景中均获得了最优的预测性能(图3)。特别的,现有工具在Zero-shot场景下均无预测能力,表明现有计算工具无法对于免疫系统未见的新肽段进行TCR亲和力识别。进而,研究团队进一步证明了PanPep可以有效地应用于:(1)T细胞克隆扩增的定量识别;(2)肿瘤新生抗原刺激下的T细胞识别;(3)新冠病毒的抗原-TCR识别。实验表明PanPep在肿瘤新生抗原预测、突发病毒的免疫学机制研究,抗原抗体设计、TIL细胞疗法等诸多领域具有广泛的应用价值。
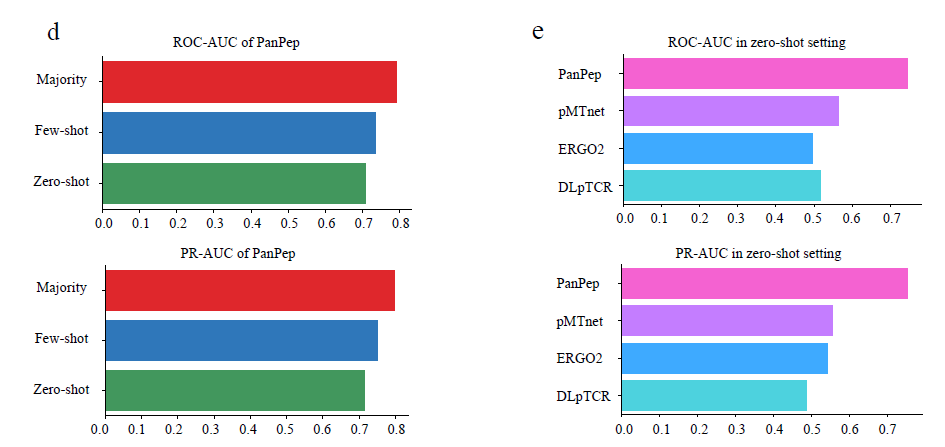
图3.PanPep性能比较
元学习和目前领域内流行的大模型(如ChatGPT等)均是通往通用人工智能(AGI,Artificial General Intelligence)的可能有效途径。本工作是应用和发展元学习理论解决生物组学数据长尾分布和小样本问题的有益尝试,是“AI for Life Science”的一个典型成功案例。该工作获得了审稿人的高度评价,评审认为:“conceptually very sound and a major algorithm advance”“motivate the development of meta learning in bioinformatics”。该工作被Nature Machine Intelligence Highlight, 并将当期特邀密苏里大学计算机科学系前系主任,美国科学促进会(AAAS)会士和美国医学和生物工程研究院(AIMBE)会士Dong Xu教授撰写Highlight View:“Meta-learning for T cell-receptor binding specificity and beyond”。在该View中,Dong Xu教授绘制了一个全面系统的框架图,清晰总结了PanPep计算模型的基本思路,并指出了生物数据中普遍存在的长尾分布特征和本工作在解决长尾分布和小样本学习上的重要价值(图4)。Dong Xu教授认为“PanPep provides a pioneering example of using meta-learning”“delivered a great promise of using meta learning to address bioinformatics' long tail distribution problems”。
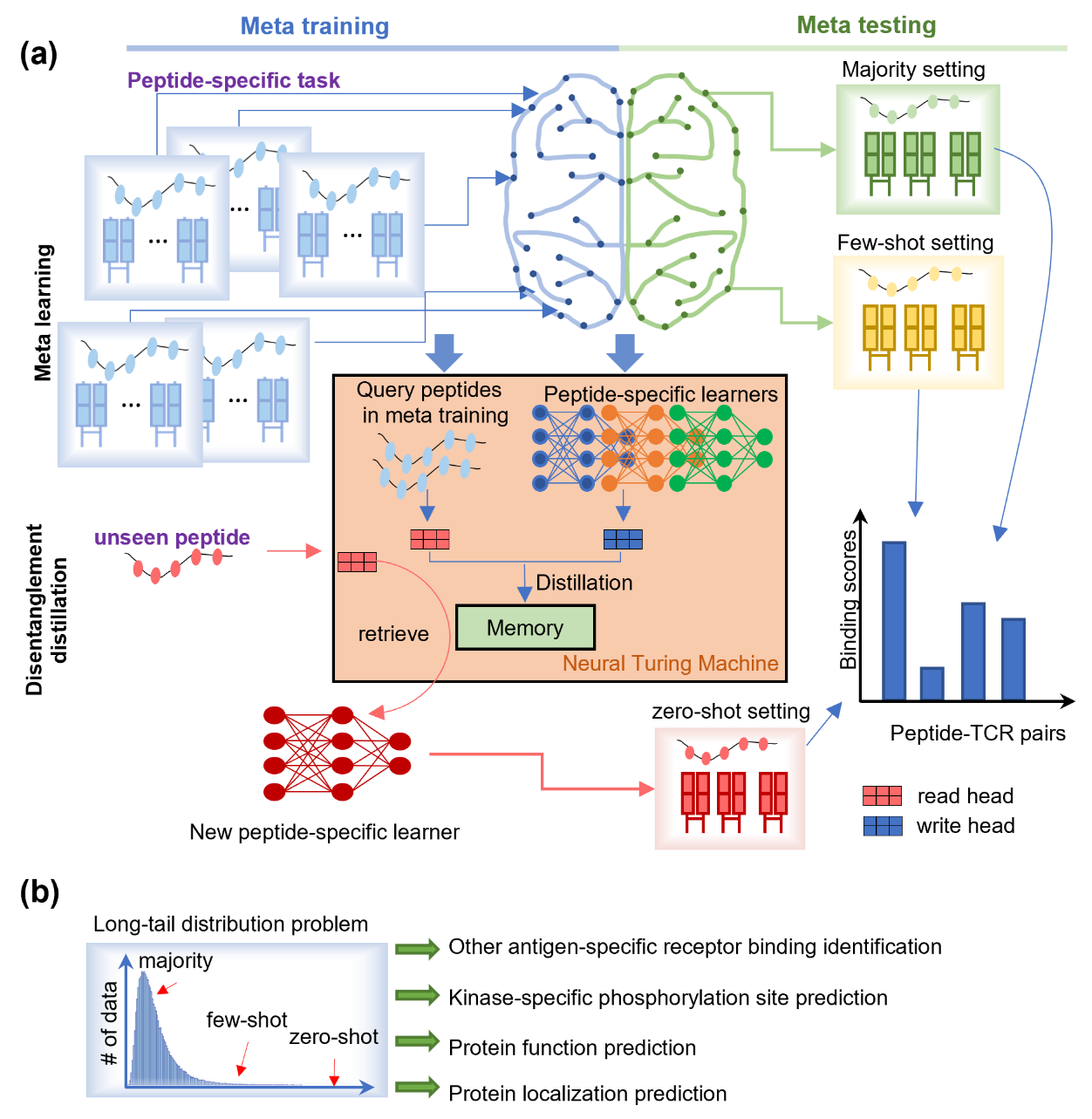
图4.PanPep框架图和生物数据的长尾分布
刘琦教授课题组长期致力于组学人工智能驱动的精准医学研究和转化实践。该论文第一作者是刘琦教授课题组的高溢骋、高雨莉博士,通讯作者是刘琦教授。该项目受到国家自然科学基金、上海市人工智能科技重大专项以及国家重点研发计划BT&IT专项资助。